最简单的质粒图谱查询方法详解(图)
1.NCBI(需要Vector软件)
网址:http://www.ncbi.nlm.nih.gov/
NCBI拥有着来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起的数据库,可以轻松查询到所需的质粒信息。以pRS303为例,具体操作如下,search下拉框中选中Nucleotide,搜索栏输入质粒名称pRS303,
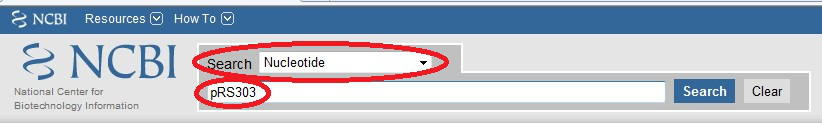
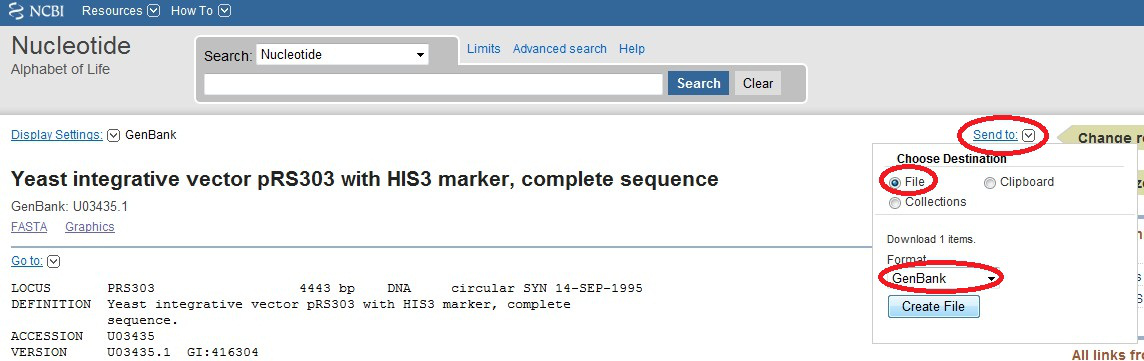
得到搜索结果,点击右边的send to,选中file,genebank等选项,点击Create File选项,得到一个gb格式的文件,再将该文件导入vector软件中,即可获得所需的质粒图谱了。
2.Vector Database
网址:https://www.addgene.org/vector-database/
Addgenen是一个全球科学家质粒共享非盈利组织。它作为一个公益性组织,负责保存和提供质粒。科学家发表文章如果涉及到质粒,会发一份到Addgene保存,其他科学家如果需要这个质粒,也可以向Addgene索取。以pRS为例,具体操作如下,在Search vector database的搜索框内输入pRS,
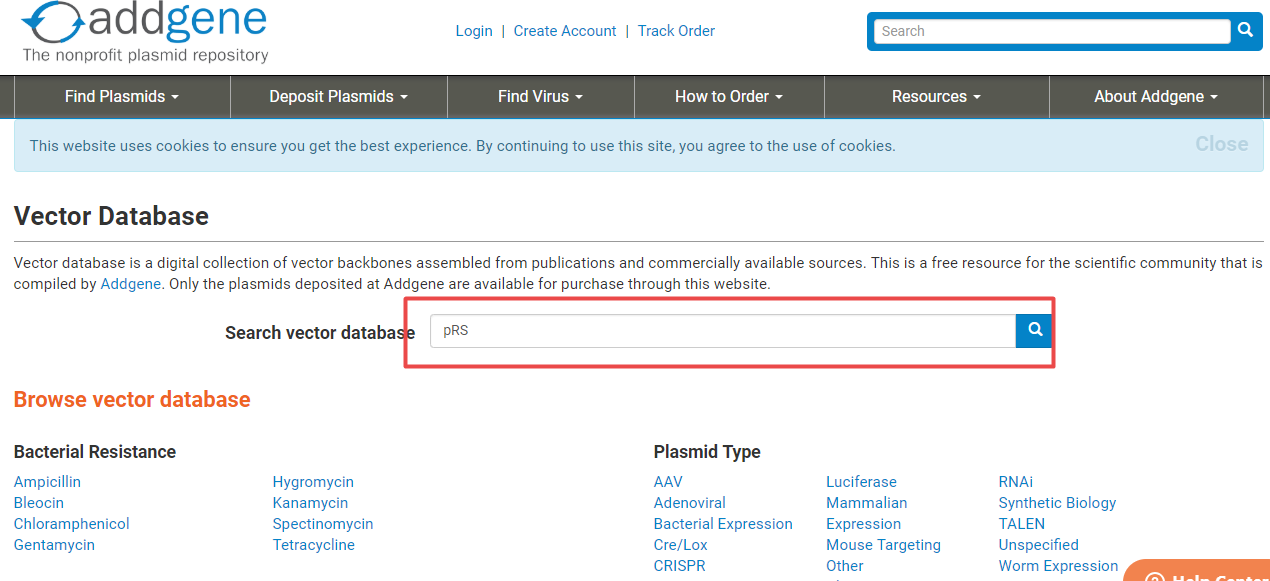
搜索结果显示了数据库中的所有pRS质粒的信息,菜单栏中分别注明了:
Name(质粒名称),Vector Type(质粒类型),Resistance Marker(抗药性标记),Bacterial Resistance(细菌抗药性),Source(来源),Sequence(质粒的序列)。
而最右侧的sequence available中, 表示此质粒没有序列,
表示此质粒没有序列, 表示可以查看详细序列。
表示可以查看详细序列。
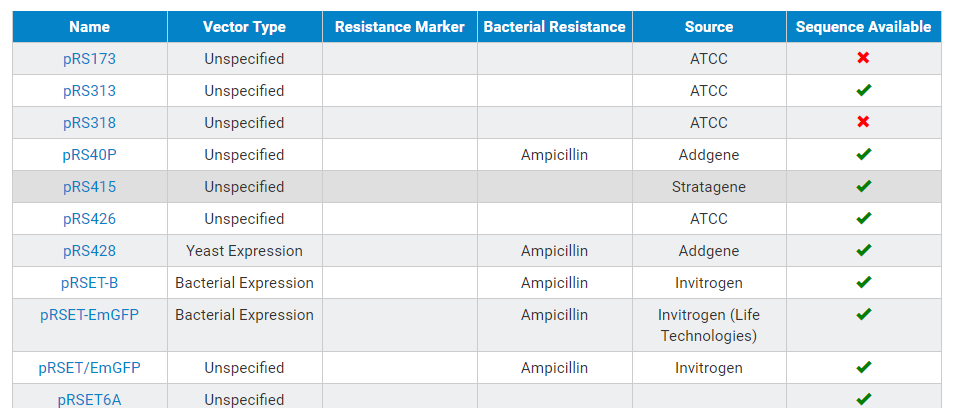
查找到自己需要的序列后,点击进入该质粒的详细介绍页(以pRSII324为例),点击View all sequences,
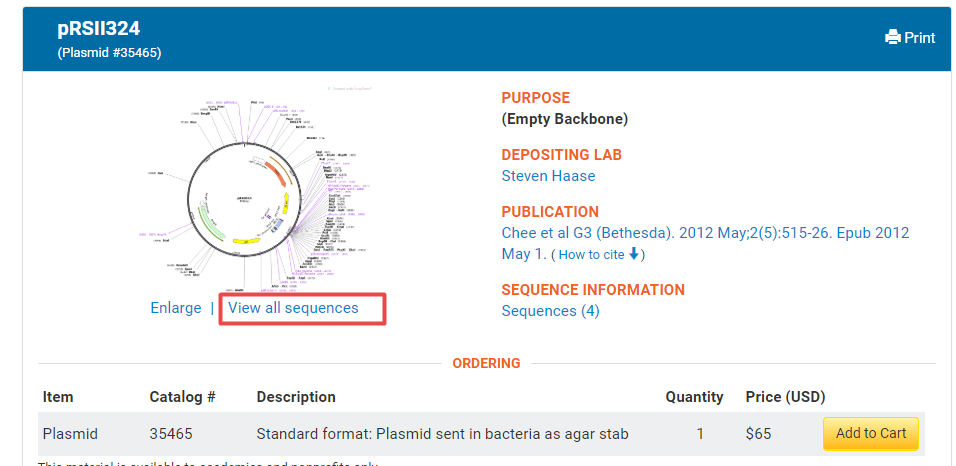
在详情页中下载GeneBank,之后导入到vector,即可查看该质粒图谱了。

3.查找改造过的质粒的质粒图谱(需要VPN)
网址:http://scholar.google.com/
有些质粒是经过改造的,所以通过上述方法不能查询到相应信息。建议在google scholar中输入质粒名称,可以直观地看哪些学者在何文章中使用了该质粒,从而可了解到质粒的来源;或者籍此向作者咨询或索取。 另外介绍下如何通过文献查找经过改造的质粒的图谱信息,
Kuhlman, T. E. and E. C. Cox (2010). "Site-specific chromosomal integration of large synthetic constructs." Nucleic Acids Res 38(6): e92.
这篇文献,介绍了一种大肠杆菌染色体整合的方法,文中介绍了三个质粒,是由作者自己改造了,如何查找到这三个质粒图谱了呢?
首先在PubMed中搜索到该文献,如下图。
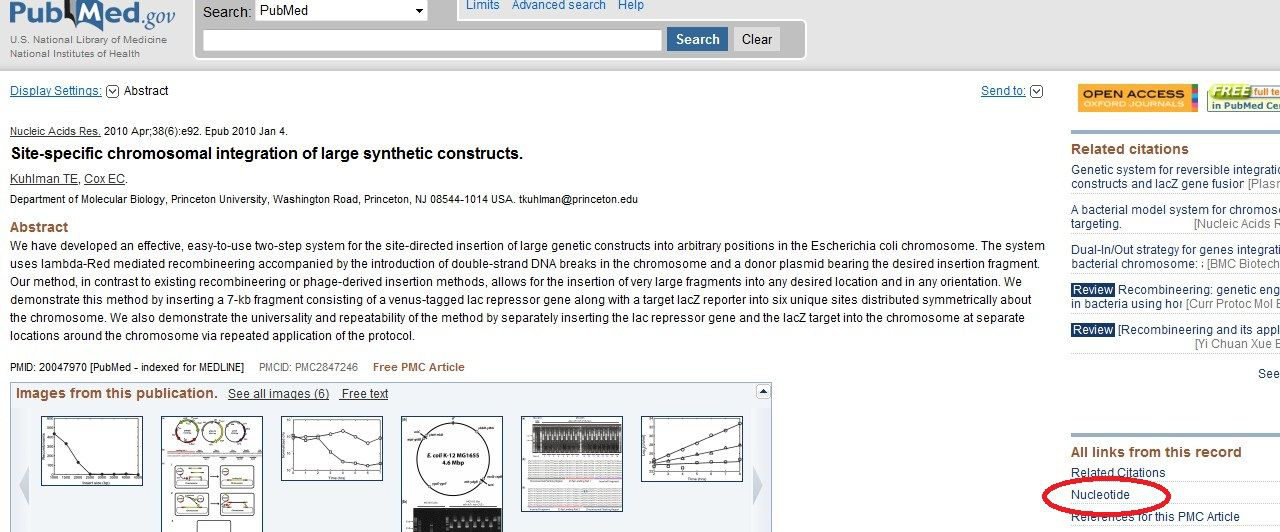
点击右边的Nucleotide,进入如下页面,看,文中涉及到的六个质粒序列全都有,点击进入,按照NCBI中下载质粒图谱的方法就可以得到这几个质粒的图谱。(该文章的作者必须已将载体信息上传了NCBI)
